基于Mamba架构的,生物医学文本分析大模型
添加书签
专注AIGC领域的专业社区,关注微软&OpenAI、百度文心一言、讯飞星火等大语言模型(LLM)的发展和应用落地,聚焦LLM的市场研究和AIGC开发者生态,欢迎关注!
生物医学文献中充斥着大量的专业术语、缩写词以及特定的语义和语法结构,这些对于传统模型来说很难处理。
例如,生物医学领域中的术语常常具有多义性,同一个词在不同的语境中可能有完全不同的含义。传统模型可能会因为无法准确把握这些术语的具体含义而产生误解,从而导致对文献内容的理解出现偏差。
生物医学领域的知识更新换代非常快,新的术语和概念不断涌现。传统模型可能无法及时跟上这些变化,导致在处理最新的生物医学文献时显得力不从心。
为了更好地分析生物医学文本,美国伦斯勒理工学院和斯坦福大学医学院的研究人员,联合开发了专业分析大模型——BioMamb。

BioMamb基于Mamba架构开发而成,可有效克服传统Transformer架构模型在处理长序列时遇到的计算效率瓶颈的难题。
传统的Transformer模型虽然在许多任务上表现出色,但由于其时间复杂度随序列长度呈二次增长,因此在处理较长文本时效率较低。
而Mamba通过采用结构化状态空间模型,将参数作为输入的函数,实现了线性的时间复杂度,从而大大提高了处理长序列数据的能力。这种特性对于生物医学文献尤为重要,因为这些文献往往包含大量的专业术语和复杂的句子结构,需要模型能够捕捉到更长范围内的上下文信息。
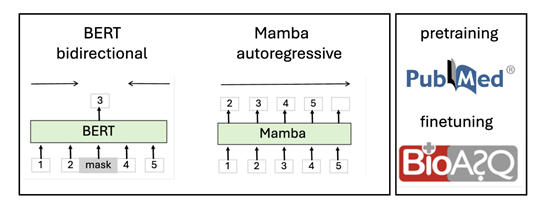
BioMamba的开发过程包括预训练和微调两个阶段:在预训练阶段,BioMamba 首先使用 Mamba – 130m 模型的权重进行初始化,然后在包括 PubMed 摘要在内的大量生物医学文本语料库上进行进一步训练。
PubMed是一个免费且非常全面的数据库,主要用于获取生命科学和生物医学主题的参考文献和摘要,是研究人员、医疗保健专业人员和学生的重要资源。
尽管Mamba 模型在初始训练时可能会遇到一些生物医学数据,但这些数据的比例通常非常小。因此,在针对生物医学语料库进行进一步预训练对于增强模型捕捉领域特定模式和术语的能力至关重要。
此外,BioMamba采用了自回归的预训练方法,通过预测给定上下文中下一个单词的方式来学习语言模型。这种方法允许模型从左至右地理解文本流,非常适合于生成连续且连贯的文本内容。
在微调阶段,BioMamba 在特定的下游任务上进行监督学习,以调整模型的参数来优化任务性能。在本研究中,重点关注的是问答(QA)任务。为了进行实验,研究人员使用了 BioASQ 事实数据集,该数据集被重新格式化为与斯坦福问答数据集(SQuAD)的结构相匹配。
在微调过程中,BioMamba 在 BioASQ 数据集上进行训练,该数据集包含生物医学 QA 对。通过微调,BioMamba 能够利用其预训练的知识并适应生物医学问答的特定要求,从而有效地解决生物医学语言的细微差别并提供准确的答案。
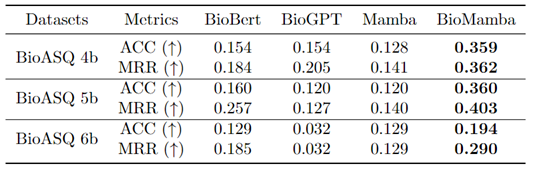
研究人员对BioMamba模型进行了广泛的测试和评估,使用了多个生物医学NLP任务的数据集,包括问答、文本分类和实体识别等。
实验结果显示,BioMamba模型展现出了卓越的性能,其准确率、精确率等指标均显著优于现有的生物医学NLP模型。
本文素材来源BioMamba论文,如有侵权请联系删除
END


本篇文章来源于微信公众号: AIGC开放社区






